L’émergence récente d’un nouveau coronavirus (Covid-19) provoque une épidémie de pneumonie virale inhabituelle. Elle a débuté en Chine à Wuhan et est maintenant étendue dans le monde. C’est un avertissement du risque posé pour la santé publique avec l’apparition de nouveaux coronavirus (CoV). Nous nous proposons après une brève introduction de décrire les caractéristiques générales des CoV puis les maladies causées par différents CoV. Cet article aidera à comprendre la biologie et les risques potentiels des CoV qui existent non seulement chez l’homme mais aussi chez les vertébrés domestiques et sauvages.
Introduction
Les CoV peuvent infecter les voies respiratoires, gastro-intestinales, hépatiques et le système nerveux central de l’homme et des animaux. Des foyers de CoV émergents, le SRAS (syndrome respiratoire aiguë sévère) en 2002/2003 en Chine et MERS (syndrome respiratoire du moyen-orient) au Moyen-Orient en 2012 ont démontré la possibilité de transmission de l’animal à l’Homme et d’homme à homme. Une flambée de pneumonie mystérieuse à Wuhan depuis décembre 2019 attire énormément l’attention. L’agent causal de cette mystérieuse pneumonie a été identifié comme un nouveau coronavirus par séquençage et enquêtes étiologiques. Le 12 janvier 2020, l’Organisation mondiale de la santé (OMS) a temporairement nommé le nouveau coronavirus SRAS-CoV2.
L’émergence sporadique et les flambées de nouveaux types de CoV nous rappellent que les CoV sont une grave menace pour la santé mondiale. Ces nouvelles flambées sont inévitables à l’avenir en raison des changements climatiques et écologiques et donc des changements d’habitats de la faune sauvage. Il est donc urgent de développer des thérapies et vaccins contre les CoV.
Structure des coronavirus
Le virus est constituée d’une capside de structure hélicoïdale qui comprend l’ARN génomique associé à la protéine N. Elle est entourée d’une enveloppe constituée des protéine E et M, de projections de la glycoprotéine S (spicule) et d’une hémagglutinine-estérase (Figure ci-dessous).
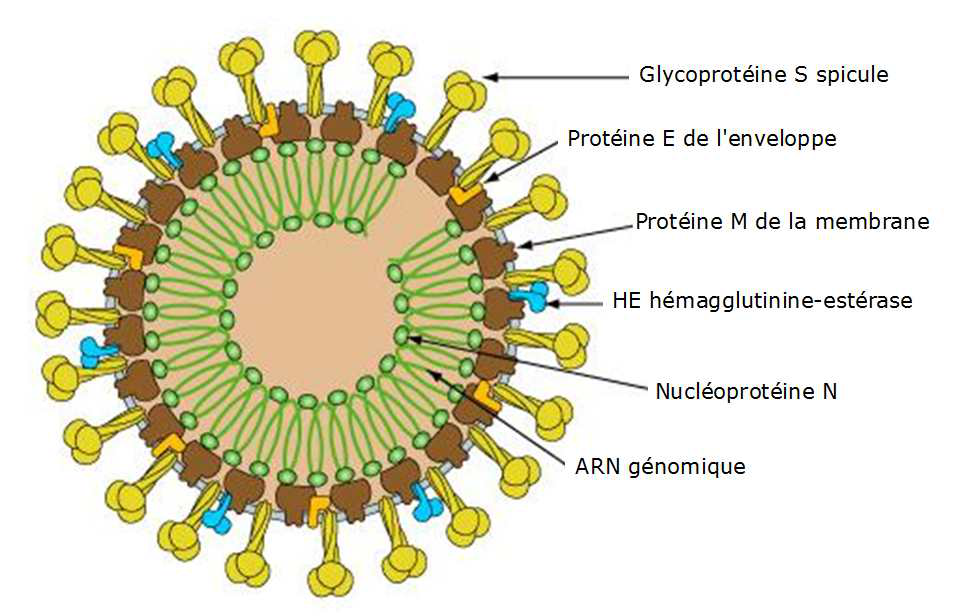
La reconnaissance d’une cellule cible est spécifique. Une protéine du virus, S, pour les CoV ou un motif particulier interagit avec une protéine ou un motif glycannique présent sur une protéine de la membrane cellulaire. Il s’ensuit une modification structurale de la membrane qui permet à une autre protéine du virus d’interagir avec un co-récepteur cellulaire et l’entrée du virus. Pour le SARS-CoV-2, la protéine S par son extrémité C-terminale reconnait comme récepteur la protéine hACE2. La protéine hACE2 est l’enzyme de conversion (isomérase) de l’angiotensine de sa forme inactive à sa forme active.
Structure du génome des coronavirus et leur réplication
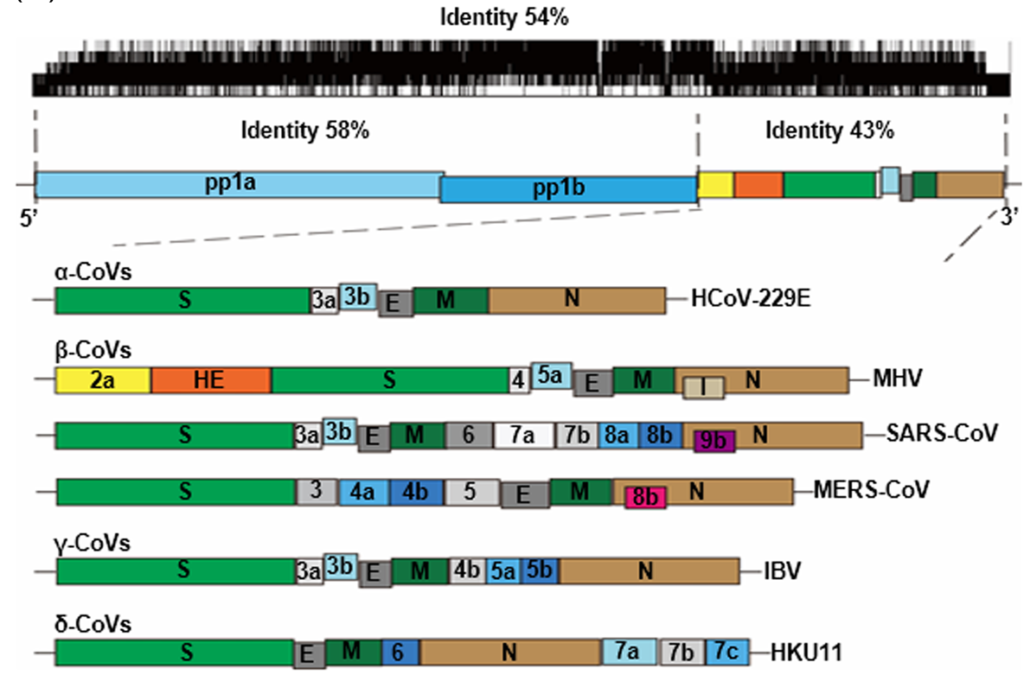
Les CoV appartiennent à la famille des Coronaviridae, sous-famille des Coronavirinae, de l’ordre Nidovirales comprend quatre genres : Alphacoronavirus, Betacoronavirus, Gammacoronavirus et Deltacoronavirus. Le SARS-CoV-2 est un betacoronavirus. Le génome des CoV est un ARN simple brin de sens positif (+ ARNsb) (~ 30 kb) avec une coiffe en 5 ‘et une queue polyA en 3’. L’ARN génomique est utilisé comme ARN messager pour être directement traduit en deux polyprotéines. Celles-ci donnent naissance à des protéines non structurales (nsp) qui forment le complexe de réplication-transcription (CRT) dans des vésicules à double membrane. Par la suite, le CRT synthétise un ensemble d’ARN génomique et sous-génomiques de polarité négative qui servent à la synthèse des différents ARN messagers. Ces ARN messagers sous-génomiques possèdent des séquences 5′-leader et 3′-terminales communes. Le génome et les sous-génomes d’un CoV typique contiennent au moins six phases ouvertes de lecture (Open reading frame, ORF). Les premières ORF (ORF1a/b), environ les deux tiers du génome, encodent 16 nsp (nsp1-16). Un décalage du cadre de lecture de -1 entre ORF1a et ORF1ab conduit à la production de deux polypeptides: pp1a et pp1ab clivés en 16 nsp. Ce clivage est réalisé par une protéase de type chymotrypsine d’origine virale dénommée protéase principale et une ou deux protéases de type papaïne. Les autres ORF sur le tiers du génome près de l’extrémité 3′ codent au moins quatre protéines. Outre ces quatre protéines structurales principales, les différents CoV codent des protéines accessoires, telle que la protéine hémagglutinine-estérase (HE). Toutes les protéines de structure et accessoires sont traduites à partir des ARN sous-génomiques des CoV.
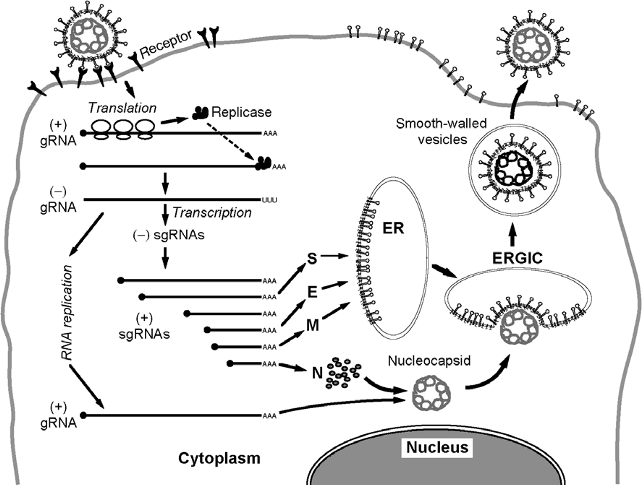
Les résultats des alignements de séquences suggèrent que les protéines non structurales (58% d’identité) sont plus conservées que les protéines structurales (43%). Ce résultat indique une probable adaptation des CoV à différents hôtes. Étant donné que les taux de mutation dans la réplication des virus à ARN sont beaucoup plus élevés que celui des virus à ADN, les génomes des virus à ARN sont généralement d’une longueur inférieure à 20 000 nucléotides (20 kb). Cependant, les génomes des CoV ont environ 30 kb de longueur, ce sont les plus grands virus à ARN connus. Le maintien d’un si grand génome chez les CoV peut être lié à la caractéristique spéciale de leur complexe de réplication qui contient plusieurs enzymes de modification des ARN parmi lesquelles une exoribonucléase 3′-5 ′ qui fournit probablement une fonction de relecture des ARN en cours de synthèse.
Fonctions des protéines structurales et non-structurales dans la réplication des coronavirus
Plusieurs des protéines non-structurales sont impliquées dans la réplication de l’ARN des CoV. Cependant, les fonctions de certaines de ces protéines sont inconnues ou mal comprises. Parmi les fonctions connues citons nsp1 qui dégrade les ARN messagers cellulaires, nsp4 et 6 qui participent à la formation de la vacuole de réplication, nsp13 une hélicase, nsp5 la protéase principale. La protéine nsp12 est la réplicase (ARN polymérase ARN dépendant et résulte du changement de phase lors de la synthèse des polypeptides pp1a et pp1ab. Les quatre protéines structurales sont essentielles pour l’assemblage des virus et l’infection des CoV. Les homotrimères des protéines S constituent les spicules de la surface du virus. La protéine M a trois domaines transmembranaires, elle favorise la courbure de la membrane et se lie à la nucléocapside. La protéine E joue un rôle dans l’assemblage du virus et est impliquée dans la pathogenèse virale. La protéine N contient deux domaines qui se lient au génome du virus via différents mécanismes. Il est rapporté que la protéine N peut se lier à nsp3 pour faciliter l’attachement du génome au CRT et favoriser l’encapsidation du génome. La protéine N est antagoniste de l’interféron (IFN) et répresseur de l’interférence à ARN qui semble être bénéfique pour la réplication virale.
Diversité de la pathogenèse des CoV
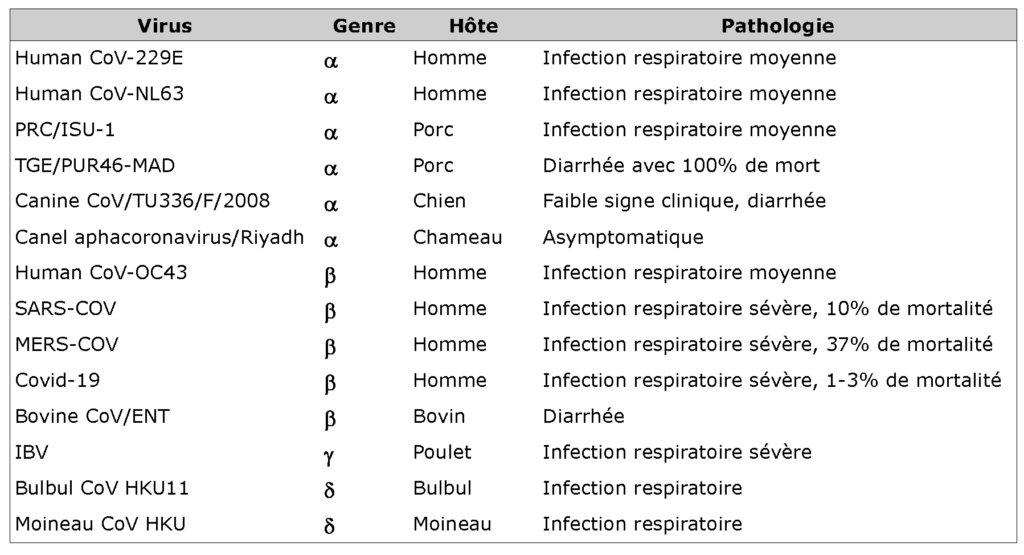
Différents CoV présentent une gamme d’hôtes variée et un tropisme tissulaire. Habituellement, les alphacoronavirus et les bétacoronavirus infectent les mammifères. En revanche, les gammacoronavirus et les deltacoronavirus infectent les oiseaux et les poissons, mais certains d’entre eux peuvent également infecter les mammifères. Avant 2019, il n’y avait que six CoV connus pour infecter l’homme et causer des maladies respiratoires : légères et dans de rares cas une infection grave chez les nourrissons, les jeunes enfants et les personnes âgées (HCoV‐229E, HCoV‐OC43, HCoV‐NL63 et HKU1) ; sévères (SRAS-CoV et le MERS-CoV). Les CoV peuvent aussi infecter le bétail, les oiseaux, les chauves-souris, les souris, les baleines et bien d’autres animaux et de ce fait causer de grandes pertes économiques. Par exemple, en 2016, un CoV de chauve-souris apparenté au CoV-HKU2 a provoqué une épidémie à grande échelle de maladie mortelle chez les porcs en Chine. Ce fut le premier cas répertorié d’une transmission entre animaux qui a décimé le bétail.
Le nouveau CoV, SARS-CoV-2 peut également infecter les voies respiratoires et provoquer une pneumonie sévère chez l’homme, mais il semble que les symptômes soient plus légers que ceux du SRAS et du MERS. De nombreux patients ont eu un contact direct ou indirect avec le Marché de gros de fruits de mer de Wuhan qui serait le lieu d’origine du déclenchement de l’épidémie. Cependant, la transmission du SARS-CoV-2 du poisson à l’homme est peu probable. Il y a deux réservoirs du CoV : un réservoir primaire : la chauve-souris et un réservoir secondaire responsable de la transmission à l’homme, le pangolin. Le pangolin est un mammifère à écailles (Pholidotes) d’Asie et d’Afrique. Le passage par un hôte intermédiaire entre la chauve-souris et l’homme permet une sélection de variants, principalement pour les protéines reconnaissant les récepteurs cellulaires, ce qui favorise le franchissement de la barrière d’espèce. En raison de la possibilité de transmission de l’animal à l’homme, les CoV du bétail et d’autres animaux, y compris les chauves-souris et les animaux sauvages vendus sur le marché doivent être surveillés en permanence. De plus, des preuves indiquent que le nouveau virus SARS-CoV-2 se propage par la voie de transmission interhumaine. Pour une meilleure compréhension de la pathogenèse des CoVs, les principaux CoVs sont répertoriés dans le tableau ci-dessous.
Traitement et prévention
À l’heure actuelle, il n’existe pas de traitement antiviral spécifique unique pour le CoV et les principaux traitements sont en cours d’évaluation. L’interféron recombinant associé avec l’antiviral ribavirine n’a que des effets limités contre l’infection. Après les épidémies de SRAS et MERS, d’importants efforts ont été consacrés au développement de nouveaux antiviraux ciblant les protéases, la polymérase, MTases et les protéines d’entrée dans les cellules. Cependant, aucun d’entre eux ne s’est révélé efficace dans les essais cliniques. Plasma et anticorps obtenus chez les patients en convalescence ont été proposés pour une utilisation dans le traitement.
Puisqu’il n’y a pas de thérapie ou de vaccin efficace, les meilleures mesures de prévention sont l’identification de la source de l’infection, le diagnostic précoce, l’isolement, les traitements de soutien et la publication en temps opportun des informations sur les épidémies pour éviter les paniques inutiles.
Les techniques de diagnostic reposent essentiellement sur l’amplification par PCR après rétrotranscription de l’ARN viral.
Vaccins
A ce jour, diverses stratégies vaccinales ont été évaluées : utilisation de virus vivants atténués ; à base de vecteurs viraux ; les vaccins à sous-unités ; les protéines recombinantes ; les vaccins à ADN.
Au 3 novembre 2020, selon l’OMS, il y a 10 essais cliniques de vaccins en phase 3 : 3 utilisent des virus inactivés (Sinovac, Institut de Biologie de Wuhan, Institut de biologie de Pékin, (Chine)), 4 sont à base de vecteur adénoviraux (Université d’Oxford-AtraZeneca (GB), Janssen (Belgique), Institut de biotechnologie de Pékin (Chine), Institut de Recherche Gamaleya (Russie)), 2 ont recours à des ARN messagers (Moderna (USA), Pfizer (USA) et BioNtech (Allemagne)) et une protéine virale (Novavax (USA)). 37 autres candidats vaccins sont en phase 1 ou 2 d’essais cliniques et 155 candidats en essais pré-cliniques.
Pour un complément d’information :
– Yu Chen, Qianyun Liu et Deyin Guo; Emerging coronaviruses : Genome stucture, replication and pathogenesis, Journal of Medical Virology, 2020, 92 : 418-423. Lire cet article
– Paul S. Masters ; The molecular biolgy of coronavirus, Advances in virus research, 2006, 66, 193-292. Lire cet article
– Sur l’utilisation de la Chloroquine voir cette synthèse : Of chloroquine and COVID-19, Franck Touret and Xavier de Lamballerie, Antiviral Research, 2020, 177, 104762. Lire cet article
Deux livres :
– Les maladies émergentes de Yazdan Yazdanpanah, QUAE éditions, 2016. Voir
– Face au risque épidémique de Didier Houssin, Edition Odile Jacob. Voir
Very valuable information Thank you for sharing
merci pour cet article
je vais beaucoup posé de question!! bonsoir
Merci pour cet post
good thanks for this