A ce jour, il n’y a pas de traitement médicamenteux efficace contre le SARS-CoV-2 contrairement à d’autres virus. Le sida (VIH) est soigné mais non guéri par une trithérapie dirigée contre la rétro polymérase et deux protéases. Il n’y a pas d’inhibiteurs de la réplication virale comme pour les virus de la grippe ou de l’herpès. Cette absence est sans doute liée à la présence d’une exonucléase 3′-5′ qui permet de corriger les erreurs de copie de l’ARN du SARS-CoV-2. Les anti-protéases virales, utilisées dans d’autres infections virales (VIH, Ebola), ne sont pas efficientes pour inhiber la formation de nouveaux virus.
La seule piste spécifique actuelle est l’utilisation soit de sérum de patients, soit des anticorps monoclonaux. À partir de sérum de patients, plusieurs équipes ont identifié les épitopes* (* voir glossaire) les plus réactifs de la protéine S (spicule) présente sur l’enveloppe virale. Cette dernière protéine est responsable de l’attachement du virus au récepteur cellulaire puis de la fusion des membranes, permettant l’entrée du virus dans la cellule. De nombreux sites O- et N-glycosylation sont présents sur cette protéine, au moins 9 sites d’O-glycosylation et 17 de N-glycosylation possèdent au moins un glucide.
L’approche la plus prometteuse pour éradiquer ce virus est l’obtention de vaccins mais aussi d’une vaccination d’une fraction importante de la population mondiale (70-80% uniformément répartie). Selon un bilan de l’OMS, au 24 décembre 2020, il y a 172 candidats vaccins dont 61 en essais cliniques Parmi ceux-ci 11 sont au moins en phase 3*. Le tableau 1 présente les différents types de candidats vaccins dont ceux en cours d’essais thérapeutiques (phases cliniques) et/ou en cours d’utilisation après une autorisation de mise sur le marché donnée par agence sanitaire comme la FA aux États-Unis.
Les différents vaccins
De nombreux candidats vaccins sont développés, ils reposent sur des approches très différentes. Les plus traditionnels sont des virus atténués ou inactivés, la deuxième approche est celle de la production de la protéine S in vitro, la troisième est celle des vecteurs viraux défectifs ou non avec comme transgène codant la protéine S et la plus récente celle des ARN messagers.
Les virus atténués ou inactivés
La variolisation a précédé la vaccination, elle reposait sur l’existence d’un variant de la variole* qui était moins mortel que la souche principale. On attribue à Lady Mary Wortley Montagu, épouse de l’ambassadeur d’Angleterre à Istambul, un rôle décisif dans l’introduction de la variolisation en Europe occidentale ; technique, d’inoculation d’homme à homme, utilisée en Chine depuis le 5ème siècle.
La plus ancienne technique de vaccination est celle des virus atténués découverte en 1796 par Edward Jenner (1749-1823). Ayant observé que les fermiers étaient protégés de la variole, il eut l’idée d’utiliser les pustules de la variole des vaches (cow pox) (ou vaccine) chez l’homme. Après

instillation de la vaccine, il fit un challenge avec un variant de la variole, utilisé dans la technique de variolisation. Il n’existe pas toujours de souche de virus atténué mais il est souvent possible d’en sélectionner comme pour le vaccin contre la rougeole. Une technique pour obtenir une souche de virus atténué est de sélectionné des mutants thermosensibles par multiplication à base température (30-33°C) puis vérifier leur non multiplication à 37°C.
Une autre possibilité d’obtention de vaccin est celle des virus inactivés. En1885, Louis Pasteur (1822-1895) a utilisé des virus dont la virulence a été atténuée pour lutter contre la rage. Pour l’inactivation, plusieurs techniques sont possibles comme un traitement par les UV, par chauffage ou chimique par exemple avec la formaldéhyde.
Dans ces deux approches, il s’agit de réduire le titre infectieux des virus tout en laissant la structure des protéines du virus, les futurs antigènes, la plus intacte.
En ce qui concerne les vaccins inactivés anti-SARS-CoV-2, ils sont principalement développés par des sociétés de biotechnologies chinoises comme : Sinovac Research and Development, Sinopharm en partenariat avec l’Institut de biologie de Wuhan ou de Pékin. Trois candidats vaccins ont été testés en phase 3 et sont maintenant utilisés en Chine.
Protéines virales
Quand la protéine virale responsable de la réponse immune est identifiée, elle est utilisée comme source d’antigène. Des cultures de cellules in vitro permettent l’obtention de ces protéines :
– les cellules CHO (cellules ovariennes d’hamster chinois), les modifications post-traductionnelles sont celles présentes chez tous les mammifères notamment les glycosylations, c’est le choix de Anhui Zhifel Longcom Biopharceutical (Chine) ;
– le système baculovirus* – cellules d’insecte Sf9 – est sans doute le plus efficient mais les modifications ne sont pas toutes identiques même si la conformation des protéines est conservée. C’est ce système qui a été choisi par le regroupement Sanofi – GSK dans le projet. Novavax.
Ces systèmes nécessitent la purification, par des techniques classiques de biochimie des protéines d’intérêt, la protéine S et/ou M, correspondant à la spicule et/ou à la matrice du SARS-CoV-2.
Les vecteurs viraux
Il existe deux types de vecteurs viraux : les virus défectifs qui ne se multiplient pas après injection, les plus utilisés dérivent des adénovirus et les virus réplicatifs, qui se multiplient après injection, c’est principalement le cas du virus de la rougeole.
Vecteurs défectifs dérivés d’adénovirus
Les adénovirus sont des virus à ADN, responsable d’infection respiratoire bénigne et pour 2 sérotypes humain (hAd40 et hAd41) de gastro-entérites chez l’enfant. Ils ont été utilisés comme vecteurs de gène en thérapie génique. Cette dernière utilisation a entraîné un développement important non seulement de leurs biologies mais aussi de nombreux outils de modifications de leurs génomes, Les plus importantes concernent la délétion de trois régions d’expression précoce (avant la réplication de l’ADN viral) : la région E3 qui permet au virus d’échapper au système immunitaire et dispensable pour la production de virus en cellules, les régions E1A et E1B indispensables à la réplication du virus dans des cellules au repos. L’absence de ces deux dernières régions rend le virus défectif à la réplication. La production de ces virus défectifs est possible par l’utilisation de cellules exprimant les protéines de ces régions c’est notamment le cas de la lignée HEK293 (human embryonic kidney cell, obtenue en 1977 par Franck Graham).
Pour les vecteurs adénoviraux, le gène d’intérêt est inséré à la place des régions E1A et E1B (voir figure), en conservant des séquences en amont qui permettent l’encapsidation du génome viral et favorise aussi l’expression, sous forme d’ARN messager du transgène, ici celui codant la protéine S.
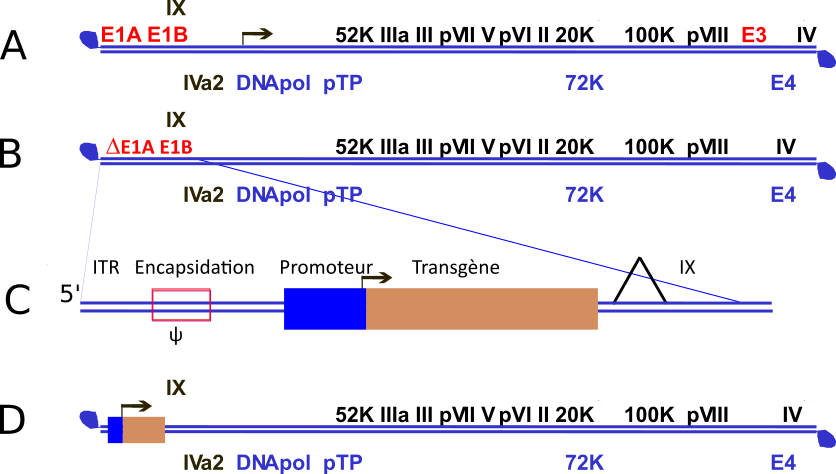
Plusieurs projets sont au moins en phase 3, seul le sérotype de l’adénovirus utilisé les distingue : Chimpanzé Ad25 pour le vaccin de l’université d’Oxford en partenariat avec AstraZenca, humain Ad26 pour Spoutnik V de Gamalya (Russie) et humain Ad5 pour Cansina (Chine).
Vecteurs réplicatifs dérivés du virus de la rougeole
Le virus de la rougeoles est un virus enveloppé de la famille des paramyxsovirus à ARN simple chaîne de polarité négative. Pour permettre la synthèse des protéines virales, il doit d’abord être répliqué en ARN positif par la réplicase virale présente dans la capside*. Les vecteurs dérivent en fait d’un variant atténué du virus de la rougeole.
Vaccins à ARN messager
doi:10.1093/nar/gkr586)
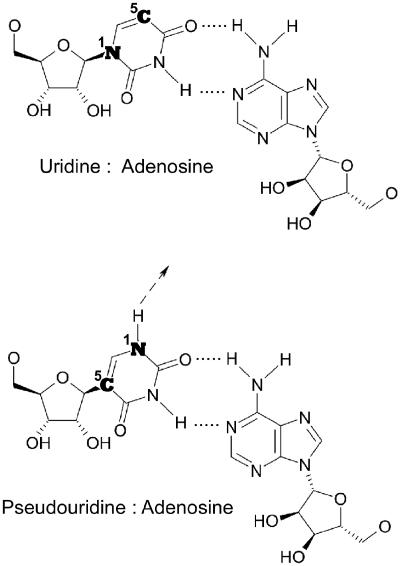
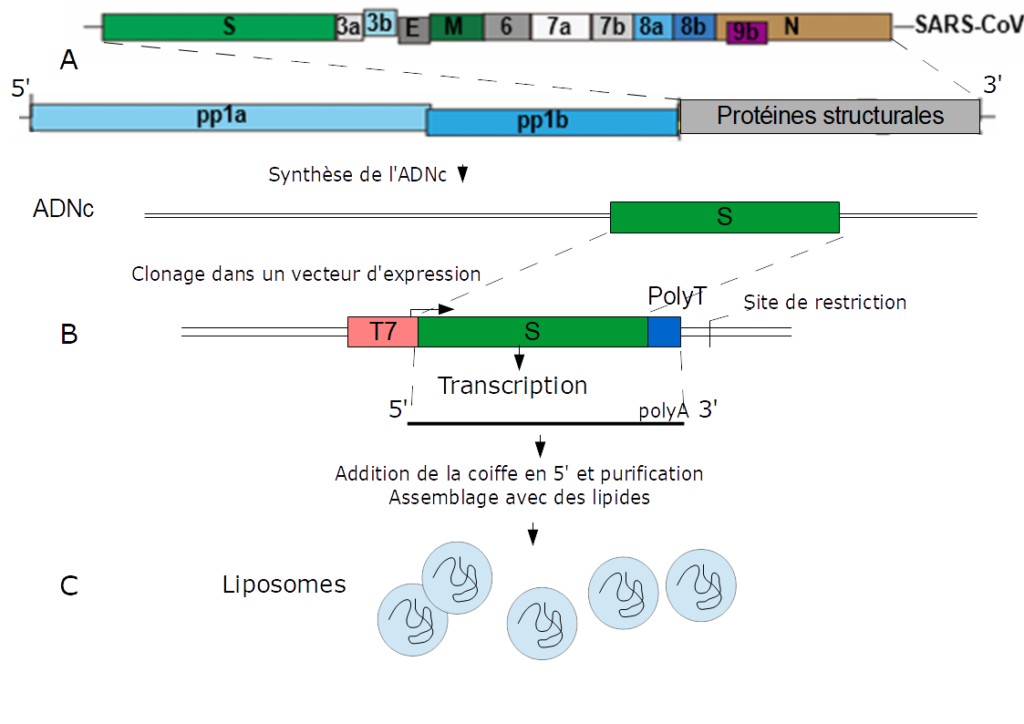
C’est à l’université de Pennsylvanie à Philadelphie, au début des années 2000, que Katalin Kariko a proposé d’utiliser l’ARN messager comme agent thérapeutique mais pour éviter une dégradation rapide elle propose l’utilisation de nucléotides modifiés. Comme son nom l’indique l’ARNm est un messager de l’information génétique, présent dans toutes les cellules, transcrit à partir de l’ADN, et traduit en protéines. Dans les cellules eucaryotes*, il possède plusieurs caractéristiques pour un fonctionnement optimal : une structure particulière à l’extrémité 5′, la coiffe* ; une séquence polyA à l’extrémité 3′ ; le codon d’initiation de la traduction, AUG, doit être dans un environnement favorable ; la phase ouverte de lecture (ORF)* se termine par un codon stop. Pour exprimer un gène étranger, il est quelquefois nécessaire de modifier les codons, en effet l’efficacité de la traduction dépend de l’abondance respective des ARNt (de transferts).

Le SARS-CoV-2 est un virus à ARN, pour l’obtention d’un vaccin, l’ARN est rétro-transcrit en ADN complémentaire (ADNc). Un fragment de cet ADNc, correspondant à la séquence codant la protéine S, de la spicule, est inséré dans un plasmide* en aval d’un promoteur reconnu par l’ARN polymérase du bactériophage T7* et en amont d’une séquence polyT (Figure ci-dessous). Après amplification du plasmide, celui-ci est linéarisé en aval de la séquence polyT, la synthèse des ARNm est réalisée in vitro par l’ARN polymérase du bactériophage T7. L’extrémité 5′ est ensuite modifiée pour obtenir une coiffe. La stabilité de l’ARNm est augmenté en remplaçant l’uridine* par une forme modifiée. L’ARNm est ensuite purifié et assemblé par des lipides dans des liposomes utilisés comme vaccins.
Pour en savoir plus
Une introduction aux vaccins :
Vaccination : agression ou protection ?
Annick Guimezanes, Marion Mathieu
Le Muscadier, Inserm
Le site de l’OMS indiquant le suivi des candidats vaccins : https://www.who.int/publications/m/item/draft-landscape-of-covid-19-candidate-vaccines
N et O-glycosylation de la protéine S : https://www.biorxiv.org/content/10.1101/2020.07.05.187344v2.full.pdf
Les résultats des essais cliniques de phase 3 du vaccin BioNTech – Pfizer
https://pubmed.ncbi.nlm.nih.gov/33301246/
Safety and Immunogenicity of Two RNA-Based Covid-19 Vaccine Candidates: https://pubmed.ncbi.nlm.nih.gov/33053279/
Glossaire
- Les eucaryotes (du grec eu, bien et karuon, noyau) regroupe tous les organismes uni- ou pluricellulaires qui se caractérisent par la présence d’un noyau dans leurs cellules.
- Un plasmide est un ADN circulaire bicaténaire capable de se répliquer de manière autonome et sans intégration dans l’ADN chromosomique d’une bactérie. Il peut posséder un gène de résistance à un antibiotique.
- ORF, open reading frame, phase ouverte de lecture : séquence d’un ARN messager qui s’étend du codon d’initiation (AUG) a un codon stop (UAG, UGA, UAA).
- Un épitope est un déterminant antigénique soit une structure moléculaire circonscrite ou un fragment d’antigène capable de déclancher une réponse immunitaire spécifique en synthétisant des anticorps ou des récepteurs de lymphocytes T.
- Le bactériophage T7 est un virus qui infecte E. coli. Les études sur ce virus ont permis des développements en biologie moléculaire et de mieux comprendre l’assemblage et l’infection virale.
- Les baculovirus sont une famille de virus en forme de batonnet spécifiques de arthropodes. Ces virus peuvent infecter plus de 600 espèces d’insectes, mais aussi de crustacés comme les crevettes. Le génome des baculovirus est constitué d’un ADN bicaténaire, circulaire, d’une taille comprise entre 80 et 180 kpb. Les baculovirus sont utilisés en biotechnologies pour la production de protéines.
All together against covid 19